データフレームの列を入れ替えて、欠損値のある行を除外する
忘れないうちに書いときます
欠損値のあるデータセットを用意します
x <- LETTERS[1:10] y <- c(1,2,NA,2,4,NA,5,1,2,5) z <- c(1,2,2,1,1,2,2,1,1,1) xyz <- data.frame(x,y,z) x y z 1 A 1 1 2 B 2 2 3 C NA 2 4 D 2 1 5 E 4 1 6 F NA 2 7 G 5 2 8 H 1 1 9 I 2 1 10 J 5 NA
Z列とY列を入れ替えます
a <- data.frame(xyz[,1]) b <- data.frame(xyz[,2]) c <- data.frame(xyz[,3]) xzy <- data.frame(a,c,b) colnames(xzy) <- c("x","z","y") x z y 1 A 1 1 2 B 2 2 3 C 2 NA 4 D 1 2 5 E 1 4 6 F 2 NA 7 G 2 5 8 H 1 1 9 I 1 2 10 J NA 5
欠損値を含む行を全て除外します
xzy_cc <- complete.cases(data.frame(xzy)) #欠損値を含む行にFALSEを返す関数・・・だからxzy_ccは完璧(complete)な行 dataset <- xzy[xzy_cc,] x z y 1 A 1 1 2 B 2 2 4 D 1 2 5 E 1 4 7 G 2 5 8 H 1 1 9 I 1 2
この処理は暗記してたがよさそうです
コピーした1行のデータをベクトルに変換
以下のようなデータをコピーしてRのベクトルに変換する方法です
10 12 14 15 18
まず以下のように入力します
x = scan()
するとコンソールに次のように表示されます
> x = scan() 1:
この1: の横に10 12 14 15 18をペーストして、enterを2回
> x = scan() 1: 10 12 14 15 18 6: Read 5 items
これでベクトルに変換できました
str(x) num [1:5] 10 12 14 15 18
ノンパラメトリック 相関係数
[,1] [,2] [,3] [1,] 27 14 5 [2,] 10 17 26 [3,] 5 12 50
上記の分割表から行の順序スコアと列の順序スコアを算出してデータセットを作成します
#RANK xr1 <- c(rep(23.5,46),rep(73,53),rep(133,67)) xc1 <- c(rep(21.5,27),rep(64,14),rep(125,5),rep(21.5,10),rep(64,17), rep(125,26),rep(21.5,5),rep(64,12),rep(125,50))
データのイメージ
xr1_xc1 <- data.frame(xr1, xc1)
相関係数を求めます
cor_r_p <- cor.test(xr1,xc1,method = "pearson") #ピアソン cor_r_s <- cor.test(xr1,xc1,method = "spearman") #スピアマン
上記の分割表から表スコアを0,1,2としてデータセットを作成します
#TABLE xr2 <- c(rep(0,46),rep(1,53),rep(2,67)) xc2 <- c(rep(0,27),rep(1,14),rep(2,5),rep(0,10),rep(1,17), rep(2,26),rep(0,5),rep(1,12),rep(2,50))
データセットにイメージ
xr2_xc2 <- data.frame(xr2, xc2)
表スコアから相関係数を求めます
cor_t_p <- cor.test(xr2,xc2,method = "pearson") #ピアソン cor_t_s <- cor.test(xr2,xc2,method = "spearman") #スピアマン
Rstudioの小ネタ (パッケージやファイルの保存方法)
Rstudioを閉じても、PC再起動してもファイルの読み込みやインストールしたパッケージは残せます (ただしパッケージは休んでいますので、起動するときにはlibraryで起こしましょう)
データ処理する前に必ず行う作業は以下の通りです
まずRstudioを起動させた状態で・・・
FileメニュからNewProject → NewProject → NewProjectと進んでいきます
Projectの名前を適当に書いてください(例:test001)、次に場所をしてしてください(as you like).
以下のように一つのフォルダの中に「---.Rhistory」と「---.Rproj」が入ってたら OK
データファイルも同じフォルダに収納します

次回から「---.Rproj」をダブルクリックしてRstudioを起動させてください
前回までに取り込んだデータは右上に表示され、そのまま使用可能です.
また右下に同じフォルダにあるデータファイルが表示されます.ここから直接データを取り込むこともできます.
またインストールしたパッケージも記憶されていますので、library( )で起動させるだけで使用できます.

Rで簡単 多重ロジスティック回帰分析
下記のサイトに移転いたしました
変数の呼称について(目的変数と説明変数)
それぞれの研究界のご意見はあると思うのですが・・・
ややこしや
目的変数  は以下のように呼ばれています
は以下のように呼ばれています
目的変数 objective variable
応答変数 response variable
反応変数 reaction variable(response variable )
結果変数 outcome variable
従属変数 dependent variable
基準変数 criterion variable
外的基準 external criterion
被説明変数 explained variable
説明変数  は以下のように呼ばれています
は以下のように呼ばれています
説明変数 explanatory variable
予測変数 predictor variable
独立変数 independent variable
パターンとしては
「目的変数&説明変数」
「従属変数&独立変数」
「被説明変数&説明変数」
個人的には…
応答変数&説明変数が理解しやすいかな?
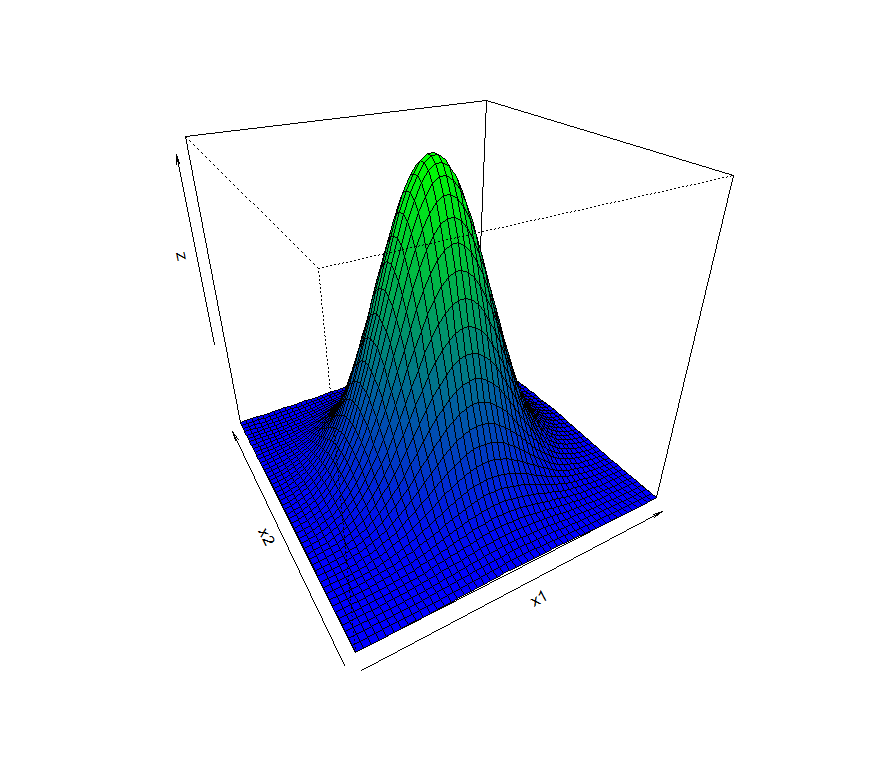
2変量の正規分布をグラフでイメージ(persp)
また、ここで勉強させていただきました.
http://cse.naro.affrc.go.jp/minaka/R/R-binormal.html
忘れないように要点のみ転記させていただます.まさに備忘録.
今回はRの関数perspを使用して、密度関数の数式から3Dのグラフを描いてみます
確率変数x1、x2が正規分布に従い、無相関であることを仮定して進めていきます.
x1 <- seq(-3, 3, length=50) # -3~3の範囲を50分割 head(x1) #先頭部分だけ確認してみます [1] -3.000000 -2.877551 -2.755102 -2.632653 -2.510204 -2.387755 x2 <- x1
これで変数の設定は完了です.次に、分散1共分散0のマトリックスを作成します.
sigma.zero <- matrix(c(1,0,0,1), ncol=2) [,1] [,2] [1,] 1 0 [2,] 0 1
Rの関数functionにx1、x2の同時確率密度を定義します. 上記sigma.zeroを分散共分散行列とする密度関数です
f <- function(x1,x2) { dmvnorm(matrix(c(x1,x2), ncol=2), mean=c(0,0), sigma=sigma.zero) } #6ポイントのみ確認してみます f(-3,-3);f(0,0);f(3,3) [1] 1.964128e-05 [1] 0.1591549 [1] 1.964128e-05 f(-1,-1);f(0,0);f(1,1) [1] 0.05854983 [1] 0.1591549 [1] 0.05854983
関数outerを使用して、(x2,x2)であらわされる座標に対しFUN(f <- function(x1,x2))を適用します.
つまり、(x2,x2)に該当する値(f)が定まることになり3次元の描画を可能にします.
z <- outer(x1, x2, f) #50×50=2500個のデータが生成されました(length(z)で確認) #10個だけ見てみましょう z[1:10] [1] 1.964128e-05 2.814820e-05 3.973927e-05 5.526846e-05 [5] 7.572219e-05 1.022015e-04 1.358875e-04 1.779878e-04 [9] 2.296621e-04 2.919285e-04
http://cse.naro.affrc.go.jp/minaka/R/R-binormal.html に書かれあります,
z の欠損値の置換は省略しております.
色塗りはRのヘルプ persp {graphics} をそのまま使用しました.
nrz <- nrow(z) ncz <- ncol(z) # Create a function interpolating colors in the range of specified colors jet.colors <- colorRampPalette( c("blue", "green") ) # Generate the desired number of colors from this palette nbcol <- 100 color <- jet.colors(nbcol) # Compute the z-value at the facet centres zfacet <- z[-1, -1] + z[-1, -ncz] + z[-nrz, -1] + z[-nrz, -ncz] # Recode facet z-values into color indices facetcol <- cut(zfacet, nbcol) persp(x1, x2, z, col = color[facetcol], phi = 30, theta = -30)