ベータ分布

- 作者: 小島寛之
- 出版社/メーカー: ダイヤモンド社
- 発売日: 2015/11/20
- メディア: 単行本(ソフトカバー)
- この商品を含むブログ (6件) を見る
この本を参考にベータ分布を勉強します.
ベータ分布:ベータ関数により導かれる分布. ベイズ推定の際に事前分布として利用します.
ベータ関数
期待値
分散
グラフを描いてみます
ベータを1に固定してαのみ大きくすると、1より大きくなると指数関数となります.
curve(dbeta(x,1,1),from = 0,to=1,ylim=c(0,5)) #黒 par(new=T) curve(dbeta(x,2,1),from = 0,to=1,ylim=c(0,5),col=2) #赤 par(new=T) curve(dbeta(x,3,1),from = 0,to=1,ylim=c(0,5),col=3) #緑 par(new=T) curve(dbeta(x,4,1),from = 0,to=1,ylim=c(0,5),col=4) #青

αとβをランダムに
curve(dbeta(x,1,1),from = 0,to=1,ylim=c(0,3)) #黒 par(new=T) curve(dbeta(x,2,2),from = 0,to=1,ylim=c(0,3),col=2) #赤 par(new=T) curve(dbeta(x,3,3),from = 0,to=1,ylim=c(0,3),col=3) #緑 par(new=T) curve(dbeta(x,5,3),from = 0,to=1,ylim=c(0,3),col=4) #青

ベータ分布の使用例1
以下は自作(架空)の問題です.
問)ある疾患の患者群で10中で3人が転倒した場合の、転倒確率をベータ分布によるベイズ統計で推測せよ.
10人中3人なのでといのが直観的な確率になるのですが、ベイズ推定では次のように考えることができます. まず転倒する確率をx、転倒しない確率を1-xとします.転倒する確率(x)+転倒しない確率(1-x)=1.xは連続無限に存在するので、確率密度となります(0≦x≦1).ここで、xの事前分布設定のために、xがどの値でも「同様に確からしい」と仮定します.つまりxの事前分布を下図のような一様分布と仮定します.

「転倒した患者3名、転倒しなかった患者7名」という結果になる確率はとなり、この数式は
のベータ分布です.
したがって転倒確率は となります.
ベータ分布の使用例2
A君は身体に障害をもっており、ただいま装具装着の練習中です.30秒以内で装着できる成功確率をxとして考えてみます.
今日は8回練習して、5回成功しました.A君の装具装着の成功率はどれくらいでしょうか? ベータ分布の使用例1と同じように、xの事前分布設定のために、xがどの値でも「同様に確からしい」と考えて一様分布を仮定します.
確率は となります.現在のA君の装具装着確率は以下のようになります.
curve(dbeta(x,6,4),from = 0,to=1,ylim=c(0,3))

事前分布が同様に確からしいということもありえないので、A君の今日の成功確率を事前分布として確率分布を推定してみます.ここでは正規化定数となるベータ関数を無視して確率分布のみを求めてみます.
P( 現状の成功確率 , 成功確率 ) = P(x) × 事前分布 =
となり以下のような確率分布(赤)となります.
curve(dbeta(x,6,4),from = 0,to=1,ylim=c(0,3)) par(new=T) curve(dbeta(x,7,4),from = 0,to=1,ylim=c(0,3),col=2)

感想)理学療法士が適切な事前分布を設定して、子どもたちの可能性を探るって重要なことだと思いました.
データフレームからの抽出 2
準備
下のデータをコピーして、Rでフレームにします
実験A 10 6 10 9 10 実験B 10 5 5 12 4 実験C 5 4 11 4 6 実験D 9 5 2 3 1
コピーして、データフレームに取り込み
(x <- read.table("clipboard",row.names = 1)) V2 V3 V4 V5 V6 実験A 10 6 10 9 10 実験B 10 5 5 12 4 実験C 5 4 11 4 6 実験D 9 5 2 3 1
列名を記載
cn <- LETTERS[1:5] colnames(x) <- cn x A B C D E 実験A 10 6 10 9 10 実験B 10 5 5 12 4 実験C 5 4 11 4 6 実験D 9 5 2 3 1
データフレームをベクトルに
準備したフレームxから2列目のみを取り出してみます
#列の取り出しは簡単です x[,2] [1] 6 5 4 5
準備したフレームxから2行目のみを取り出してベクトルにします
#2行目 x[2,] A B C D E 実験B 10 5 5 12 4 #2行目をベクトルに変換 (xv <- as.integer(x[2,])) [1] 10 5 5 12 4
マトリックスにしてベクトルとして一括抽出
(xm <- as.matrix(x)) A B C D E 実験A 10 6 10 9 10 実験B 10 5 5 12 4 実験C 5 4 11 4 6 実験D 9 5 2 3 1 (as.vector(xm)) [1] 10 10 5 9 6 5 4 5 10 5 11 2 9 12 4 3 10 4 6 1
因子ベクトル(カテゴリカルデータ)
実験A 10 6 10 9 10 a 実験B 10 5 5 12 4 a 実験C 5 4 11 4 6 b 実験D 9 5 2 3 1 b
コピーしてRのフレームに取り込みます
(xc <- read.table("clipboard",row.names = 1)) V2 V3 V4 V5 V6 V7 実験A 10 6 10 9 10 a 実験B 10 5 5 12 4 a 実験C 5 4 11 4 6 b 実験D 9 5 2 3 1 b cn2 <- LETTERS[1:6] colnames(xc) <- cn2 xc A B C D E F 実験A 10 6 10 9 10 a 実験B 10 5 5 12 4 a 実験C 5 4 11 4 6 b 実験D 9 5 2 3 1 b
上記のような場合には文字が因子に自動的に変換されています
#5列目は整数、6列目は因子 class(xc[,5]) [1] "integer" class(xc[,6]) [1] "factor"
A列、B列のF列bのみ取り出す
subset(xc,select = c(A,B),subset = F=="b") A B 実験C 5 4 実験D 9 5
A列、B列 の F列a VS b
matrixからベクトルに変換してa VS bの配列にします
(xxx <- c(as.vector(as.matrix(xca)),as.vector(as.matrix(xcb)))) xxx [1] 10 10 6 5 5 9 4 5 (dat <- array(xxx, dim = c(2,2,2))) , , 1 [,1] [,2] [1,] 10 6 [2,] 10 5 , , 2 [,1] [,2] [1,] 5 4 [2,] 9 5
項目名などを記入
name <- list("実験"=c("実験A","実験B"),"結果"=c("A","B"),F=c("a","b")) dimnames(dat)<-name dat , , F = a 結果 実験 A B 実験A 10 6 実験B 10 5 , , F = b 結果 実験 A B 実験A 5 4 実験B 9 5
共分散構造分析(パス図の描き方)
Rを使ったパス図作成の方法を忘れないうちに簡単に書いておきます
青木先生のデータを借用しまして勉強していきます.
R -- 因子分析(factanal を援用する)
dat <- matrix(c( -1.89, -0.02, 0.42, 1.23, -1.53, 0.06, 1.81, -0.59, -0.75, -0.12, 2.58, -0.20, -1.92, -0.49, -0.35, 0.69, -0.66, -0.77, -1.92, 0.38, -1.05, 0.07, -0.37, -0.89, -1.62, -2.73, 1.40, 0.18, -0.09, 0.13, 0.95, 0.84, 1.19, 1.19, 0.10, 0.93, 1.17, -1.70, 1.46, -0.25, -0.04, -0.12, -0.34, -0.24, 1.88, 0.16, 1.03, -0.05, -0.73, 0.04 ), byrow=TRUE, ncol=5) colnames(dat) <- c("v1","v2","v3","v4","v5") #列名を加えます x <- dat
相関行列、共分散行列
#後でsemパッケージのために必要になります r <- cor(x) # 相関行列 cov <- cov(x) # 共分散行列
ここはモデルをイメージするところで、色々なパターンが考えられます
RjpWikiに習って、下記のようなイメージで作業を進めて行きます.

library(sem) model <- specifyModel() v1 < F1,path1,1 v2 < F1,path2,NA v3 < F1,path3,NA v4 < F1,path4,NA v5 < F1,path5,NA v1 <> v1,s1,NA v2 <> v2,s2,NA v3 <> v3,s3,NA v4 <> v4,s4,NA v5 <> v5,s5,NA F1 <-> F1,NA,1 #終了したらCtrl+R
いよいよsemパケージを使用します
相関行列を使用したSEM
library(sem) # semパッケージ ans1 <- sem(model, r, N=10) #相関行列を使用してsemを実行 summary(ans1) Model Chisquare = 1.925355 Df = 5 Pr(>Chisq) = 0.8593742 AIC = 21.92536 BIC = -9.58757 Normalized Residuals Min. 1st Qu. Median Mean 3rd Qu. Max. -0.3976000 -0.0000002 0.0155300 0.1221000 0.2435000 0.6971000 R-square for Endogenous Variables v1 v2 v3 v4 v5 0.6297 0.0809 0.4573 0.0583 0.0506 Parameter Estimates Estimate Std Error z value Pr(>|z|) path1 0.7935558 0.4882341 1.6253592 0.10408604 v1 <--- F1 path2 -0.2843858 0.3849962 -0.7386717 0.46010637 v2 <--- F1 path3 -0.6762525 0.4525192 -1.4944171 0.13506663 v3 <--- F1 path4 -0.2414672 0.3860898 -0.6254172 0.53169727 v4 <--- F1 path5 0.2248359 0.3864879 0.5817413 0.56074096 v5 <--- F1 s1 0.3702693 0.6626860 0.5587401 0.57633909 v1 <--> v1 s2 0.9191248 0.4485423 2.0491376 0.04044867 v2 <--> v2 s3 0.5426826 0.5322169 1.0196644 0.30788765 v3 <--> v3 s4 0.9416935 0.4546109 2.0714275 0.03831887 v4 <--> v4 s5 0.9494489 0.4567576 2.0786712 0.03764758 v5 <--> v5 Iterations = 15 #Estimate の部分が非標準化係数ですが、相関行列から求めているので不正確
標準化係数
stdCoef(ans1) # 標準化係数 Std. Estimate path1 path1 0.7935557 v1 <--- F1 path2 path2 -0.2843858 v2 <--- F1 path3 path3 -0.6762525 v3 <--- F1 path4 path4 -0.2414672 v4 <--- F1 path5 path5 0.2248359 v5 <--- F1 s1 s1 0.3702693 v1 <--> v1 s2 s2 0.9191247 v2 <--> v2 s3 s3 0.5426826 v3 <--> v3 s4 s4 0.9416936 v4 <--> v4 s5 s5 0.9494488 v5 <--> v5 1.0000000 F1 <--> F1
共分散行列を使用したSEM
ans2 <- sem(model, cov, N=284) # 非標準化係数 summary(ans2) Model Chisquare = 60.54173 Df = 5 Pr(>Chisq) = 9.392241e-12 AIC = 80.54173 BIC = 32.29686 Normalized Residuals Min. 1st Qu. Median Mean 3rd Qu. Max. -2.230000 -0.000001 0.087080 0.684600 1.365000 3.909000 R-square for Endogenous Variables v1 v2 v3 v4 v5 0.6297 0.0809 0.4573 0.0583 0.0506 Parameter Estimates Estimate Std Error z value Pr(>|z|) path1 1.2135217 0.13314543 9.114257 7.921129e-20 v1 <--- F1 path2 -0.2329064 0.05622870 -4.142127 3.440994e-05 v2 <--- F1 path3 -0.6310501 0.07530436 -8.379995 5.293016e-17 v3 <--- F1 path4 -0.2643794 0.07538517 -3.507048 4.531080e-04 v4 <--- F1 path5 0.2209609 0.06773505 3.262135 1.105764e-03 v5 <--- F1 s1 0.8658805 0.27636060 3.133155 1.729383e-03 v1 <--> v1 s2 0.6164834 0.05365106 11.490610 1.470704e-30 v2 <--> v2 s3 0.4725590 0.08264694 5.717805 1.079092e-08 v3 <--> v3 s4 1.1288825 0.09718675 11.615601 3.433641e-31 v4 <--> v4 s5 0.9170030 0.07867069 11.656221 2.133019e-31 v5 <--> v5 #Estimate ここは間違いなく非標準化係数です
パス図の作成
install.packages("DiagrammeR") install.packages("scales") pathDiagram(ans1, ignore.double=FALSE, edge.labels="values", digits=3, node.font=c("HGRGE", 10)) pathDiagram(ans2, ignore.double=FALSE, edge.labels="values", digits=3, node.font=c("HGRGE", 10))


lavaanパッケージによる方法
RjpWikiより
installed.packages("lavaan") library("lavaan") model2 <- 'f1 =~ v1 + v2 + v3 + v4 + v5 ' ans3 <- cfa(model=model2, data=x, std.lv=TRUE, mimic="EQS") # mimicオプションを"EQS"とするとsemパッケージの結果と一致する。デフォルトでは"Mplus" summary(ans3, fit.measures=TRUE, standardize = TRUE) standardizedSolution(object =ans3 )# 標準化推定値 lhs op rhs est.std se z pvalue 1 f1 =~ v1 -0.794 NA NA NA 2 f1 =~ v2 0.284 NA NA NA 3 f1 =~ v3 0.676 NA NA NA 4 f1 =~ v4 0.241 NA NA NA 5 f1 =~ v5 -0.225 NA NA NA 6 f1 ~ f1 -3.576 NA NA NA 7 v1 ~~ v1 0.370 NA NA NA 8 v2 ~~ v2 0.919 NA NA NA 9 v3 ~~ v3 0.543 NA NA NA 10 v4 ~~ v4 0.942 NA NA NA 11 v5 ~~ v5 0.949 NA NA NA 12 f1 ~~ f1 1.000 NA NA NA
信頼区間のプロット
同じサイズのデータサンプルからt分布を利用した信頼区間の作図
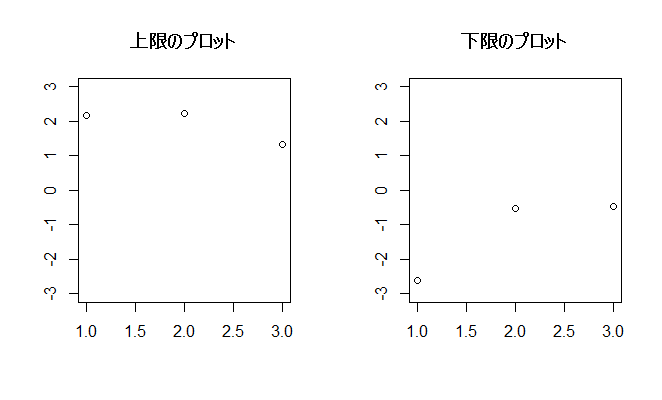
まずは3×4の場合(サンプルサイズ3を4回実施する)
x <- matrix(NA,nrow=3,ncol=4) #3×4の空セル for (i in 1:4){ #列数分乱数を代入 x[,i] <- rnorm(3) #標準正規分布の乱数を行数分繰り返す } mx <- rowMeans(x) #行平均を一括計算 mean(x[1,]) #1行目のみ取り出して確認 vx <- apply(x,1,var) #行分散を一括計算 var(x[1,]) #1行目のみ取り出して確認 lx1 <- mx+qt(0.975,2)*sqrt(vx/3) #95%信頼区間の上限(平均+t値*標準誤差), lower.tail=TRUE lx2 <- mx-qt(0.975,2)*sqrt(vx/3) #95%信頼区間の下限(平均-t値*標準誤差), (0.975,2, lower.tail=F )=qt(0.025,2,lower.tail=FALSE ) par(mfrow = c(1,2)) #1行2列に図を並べます plot(lx1, ylim = c(-3,3), main = "上限のプロット", ylab="", xlab="") plot(lx2, ylim = c(-3,3), main = "下限のプロット", ylab="", xlab="") dev.off() #作図を終了します #t値はqt(0.025,2)~qt(0.975,2)になります, lower.tail=TRUE
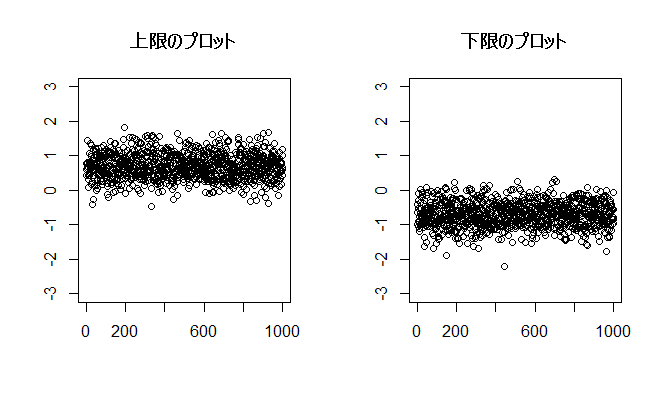
行列数値を増やしていけば見えてきます 1000行10列でやってみます
x <- matrix(NA,nrow=1000,ncol=10) for (i in 1:10){ x[,i] <- rnorm(1000) } mx <- rowMeans(x) vx <- apply(x,1,var) lx1 <- mx+qt(0.975,9)*sqrt(vx/10) lx2 <- mx-qt(0.975,9)*sqrt(vx/10) par(mfrow = c(1,2)) plot(lx1, ylim = c(-3,3), main = "上限のプロット", ylab="", xlab="") plot(lx2, ylim = c(-3,3), main = "下限のプロット", ylab="", xlab="") dev.off()
行数=r,列数=lとした場合
x <- matrix(NA,nrow=r,ncol=l) for (i in 1:l){ x[,i] <- rnorm(r) } mx <- rowMeans(x) vx <- apply(x,1,var) lx1 <- mx+qt(0.975,l-1)*sqrt(vx/l) lx2 <- mx-qt(0.975,l-1)*sqrt(vx/l) par(mfrow = c(1,2)) plot(lx1, ylim = c(-3,3), main = "上限のプロット", ylab="", xlab="") plot(lx2, ylim = c(-3,3), main = "下限のプロット", ylab="", xlab="") dev.off()
ベイズの定理でモンティ・ホール問題を考える
内容を修正しております しばらくお待ちください
LaTeXでプレゼン
無料ソフトのみで統計からプレゼンまで!
spss, word, power pointを使用しないで以下のような2枚のスライドを作ってみました.
使ったもの
- Hatena Blog
- R
- LeTeX


画像は オッズ比の信頼区間 - 統計学備忘録 since2016 からのコピペです
%LaTexの記載は以下の通りです. %タイプセットはpLaTex(ptex2pdf)を選択します \documentclass[slide,papersize]{jsarticle} \usepackage[dvipdfmx]{color} \usepackage{pxfonts} \def \sheet #1{ \section*{\centering \large \bfseries #1} } \title{\LaTeX!でプレゼンに挑戦} \usepackage[dvipdfmx]{graphicx} %図 %http://www.wankuma.com/seminar/20090912nagoya09/5.pdf \begin{document} \maketitle \begin{center} \includegraphics[width=60mm,scale=0.8]{WS000004.jpg} %絵も小さくしておきます \end{center} $p'A = \frac{a}{a+b}, p'B = \frac{c}{c+d}, \frac{\frac{p’A}{1-p’A}}{\frac{p’B}{1-p’B}}=\frac{p’A(1 - p'B)}{p’B(1 - p'A)}=\frac{\frac{a}{b}}{\frac{c}{d}}$ \end{document}
全て、以下のページに書かれてます
http://www.wankuma.com/seminar/20090912nagoya09/5.pdf
オッズ比の信頼区間
オッズ比、見込み比(odds ratio)または交差積比(cross-product)
前提
比は分母が小さくなると、数値が大きくなりすぎて正規近似の精度が悪くなります.比の対数であれば高い精度で正規近似することが可能になります. したがって、比の対数を考えていくことになります.
xm <- matrix(c("a","b","c","d"), nrow=2, byrow=T) name <- list("暴露"=c("あり(A)","なし(B)"),"疾病"=c("あり","なし")) dimnames(xm)<-name xm 疾病 暴露 あり なし あり(A) "a" "b" なし(B) "c" "d"
オッズ比=
暴露なし(A群)が、暴露あり(B群)より疾病に罹患する可能性が大きいか、小さいかを表す尺度.
p'A, p'B が小さい場合には、リスク比と同じ値になります.
オッズ比の信頼区間
比の対数を考えていきます.
標本のオッズ比=標本オッズ比( )
母集団のオッズ比を母オッズ比()とします.
標本オッズ比の対数 、 母リスク比の対数
正規近似で信頼区間を求めていくためには、以下の式が必要になります.
したがって、以下の式が95%信頼区間を求める式になります
以下、 とします
より
デルタ法(delta method)によって近似的に求めたもの(参考webより)
#Rで95%信頼区間を求めてみます rr <- a*(c+d)/(a+b)/c # = (a/(a+b))/(c/(c+d)) exp(log(rr)+c(1, -1)*qnorm ( c(0.025,0.975) )*sqrt(b/a/(a+b)+d/c/(c+d))) 例) クロス表イメージ (x <- matrix(c(3, 5, 6, 8 ),2,byrow=T)) [,1] [,2] [1,] 3 5 [2,] 6 8 a<-3; b<-5; c<-6; d<-8 #参考webより ( or <- a*d/(b*c) ) #オッズ比 [1] 0.8 or*exp(qnorm ( c(0.025, 0.975) )*sqrt( 1/a + 1/b + 1/c + 1/d) ) [1] 0.1348801 4.7449559 #Rの関数を使えば瞬間で色々出力してくれます(やっぱり凄い) install.packages("Epi") library(Epi) twoby2(x) 2 by 2 table analysis: ------------------------------------------------------ Outcome : Col 1 Comparing : Row 1 vs. Row 2 Col 1 Col 2 P(Col 1) 95% conf. interval Row 1 3 5 0.3750 0.1254 0.7152 Row 2 6 8 0.4286 0.2065 0.6837 95% conf. interval Relative Risk: 0.8750 0.2972 2.5763 #リスク比と95%信頼区間 Sample Odds Ratio: 0.8000 0.1349 4.7450 #オッズ比と95%信頼区間 Conditional MLE Odds Ratio: 0.8081 0.0884 6.3780 #Fisherの正確検定によるオッズ比と95%信頼区間 Probability difference: -0.0536 -0.3956 0.3312 Exact P-value: 1 #Fisherの正確検定によるp値 Asymptotic P-value: 0.8059 #正規近似によるp値 ------------------------------------------------------
参考web1 統計学入門−第3章
参考web2 R による統計処理
参考文献)柳川 堯 ; 観察データの多変量解析―疫学データの因果分析,近代科学社 ,2016